[R-Machine Learning] 3. kNN (k Nearest Neighbor)

1. kNN(k Nearest Neighbor)
- 범주를 알지 못하는 예제의 범주를 분류되어있는 가장 유사한 예제의 범주로 지정하는 작업
- 유사한 범주의 아이템은 수치, 동질 이다.
- 복잡하거나 이해하기 어려운 속성과 목적 범주에 관계된 분류 태스크에 적합함
1) 장점 및 단점
- 장점
- 단순하며 효율적이다.
- 데이터 분산에 대한 추청을 만들 필요가 없다.
- 빠른 훈련 단계
- 단점
- 모델을 생성하지 않는다
= 속성 사이의 관계에서 통찰력을 발견하는 능력이 제한됨 - 느린 분류
- 많은 메모리 필요
- 명목형 변수, 결측 데이터의 경우 추가 처리가 필요함
- 모델을 생성하지 않는다
- 명목형 변수인 범주로 구성될 예제 데이터 셋으로 훈련함
- 훈련 데이터에서 유사도로 가장 가까운 k개의 데이터를 찾는다.
2) 거리 계산
- 최근접 이웃을 계산하려면 거리함수 혹은 인스턴스 간의 유사도를 측정할 공식이 필요함
- 해당 예제에서는 유클리드 거리를 사용한다.
$ dist(p,q) = sqrt{ {(p_1 - q_1)}^2 + {(p_2 - q_2)}^2 + … + {(p_n - q_n)}^2 } $
3) k 값 선택
- k 값을 결정하는 것 = 모델이 미래의 데이터에 대해 얼마나 잘 일반화할지를 결정한다.
- k를 큰 값으로 설정할 경우 노이지 데이터로 변화량은 줄어들지만 중요한 패턴을 무시할 수 있다.
- 반대로 k를 작은 값으로 설정할 경우 노이지 데이터나 이상치에 영향을 받는다.
- 때문에 일반적으로 k 값은 3~10 사이의 값으로 지정한다.
2. kNN 예제
-
사용 데이터: 유방암 진찰 데이터( http://archive.ics.uci.edu/ml 의 Breast Cancer Wisconsin Diagnostic)
https://archive.ics.uci.edu/dataset/15/breast+cancer+wisconsin+original -
데이터 셋 내의 속성 변수
- 반지름
- 텍스처
- 둘레
- 면적
- 평활도
- 다짐도
- 요면
- 요면점
- 대칭
- 프렉탈 차원
- 진단 여부 : 양성(B) / 악성(M)
1) 분포 확인
[R code]
setwd("D:/workspace/R/workspace")
cancer <- read.csv("wisc_bc_data.csv")
cancer <- cancer[,-1]
table(cancer$diagnosis)
[결과]

[R code]
cancer$diagnosis <- factor(cancer$diagnosis, levels = c("B","M"),
labels = c("Benign", "Malignant"))
round(prop.table(table(cancer$diagnosis)) * 100, digits = 1)
[결과]

[R code]
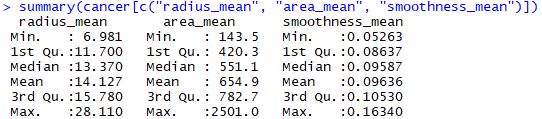
summary(cancer[c("radius_mean", "area_mean", "smoothness_mean")])
[결과]
2) 일반화
- 각 변수별로 측정범위가 다르기 때문에 값을 재조정하여 잠재적인 문제점을 해결할 수 있다.
- 각 변수를 적용하기 위해 lapply() 를 사용해 normalize 함수를 적용시킨다.
[R code]
# Normalize
normalize <- function(x) {
return((x - min(x)) / (max(x) - min(x)))
}
cancer_n <- as.data.frame(lapply(cancer[2:31], normalize))
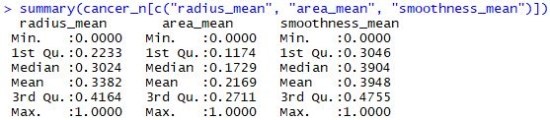
summary(cancer_n[c("radius_mean", "area_mean", "smoothness_mean")])
[결과]
- 모델이 데이터 셋에 과적합 되는 것을 막기 위해 train 데이터와 test 데이터로 나누어 학습을 진행한다.
[R code]
train <- cancer_n[1:469,]
test <- cancer_n[470:569,]
- 목표 변수인 diagnosis를 배제한다.
- kNN의 경우 훈련을 위해 훈련용 , 테스트 용 데이터 셋으로 나눈 팩터형 벡터로 범주 라벨을 저장해둬야한다.
[R code]
train_label <- cancer[1:469, 1]
test_label <- cancer[470:569, 1]
3) 모델링
- kNN은 class 패키지에 포함되어 있으며 모델에 사용할 매개 변수값들은 다음과 같다.
ex. model <- knn(train, test, class, k)
* train : 수치형 데이터로 구성된 학습용 데이터 셋
* test : 수치형 데이터로 구성된 테스트용 데이터 셋
* class : 훈련 데이터의 각 행에 대응되는 범주형 데이터로 구성된 라벨
* k : 최근접 이웃의 개수 , 정수형으로 명시
- k 값 선정 : k^2 이 학습 데이터의 행의 개수에 근접하도록 설정한다.
[R code]
# Modeling
install.packages("class")
library(class)
test_pred <- knn(train = train, test = test, cl = train_label, k = 21)
4) 모델 성능 평가
- 작성한 모델의 성능을 평가하기 위해 gmodels 패키지의 CrossTable() 함수를 사용해 교차표를 작성한다.
- 작성 시 실제 값으로는 test_label을 사용하며 카이제곱 값은 사용하지 않으므로 FALSE로 설정한다.
[R code]
# Test
install.packages("gmodels")
library(gmodels)
CrossTable(x = test_label, y = test_pred, prop.chisq = F)
[결과]
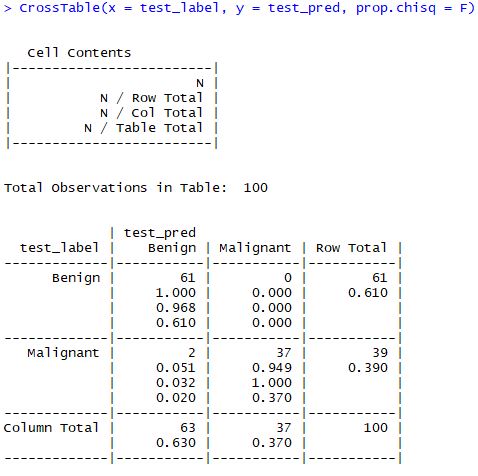
- 확인할 결과 전체 100개 중에서 98% 를 골라냈다.
5) 모델 성능 높이기
- 이전 모델에 비해서 2가지의 변화를 준다.
* z-score 표준화
* k 값의 변화
(1) z - score 표준화
- 정규화는 kNN에서 일반적으로 사용되지만 항상 속성을 축소하는데에는 적합하지 않다.
- z -score 의 경우 표준화된 값은 최소와 최대를 미리 정하지 않았기 때문에 극단적으로 값이 중앙에 모이지 않는다.
- 때문에 거리 계산에서 이상치에 좀 더 큰 가중치를 부여하는 것이 합리적이라고 판단된다.
- 해당 예제에서는 벡터 표준화를 위해 scale() 함수를 사용한다.
- 기본적으로 z-score를 사용하기 때문에 값을 축소하고, lapply를 사용하지 않고도 정규화하는 것이 가능하다.
[R code]
# Modify model
cancer_z <- as.data.frame(scale(cancer[-1]))
summary(cancer_z$area_mean)
train_z <- cancer_z[1:469,]
test_z <- cancer_z[470:569,]
test_z_pred <- knn(train_z, test_z, cl = train_label, k=21)
CrossTable(x = test_label, y = test_z_pred, prop.chisq = F)
[결과]
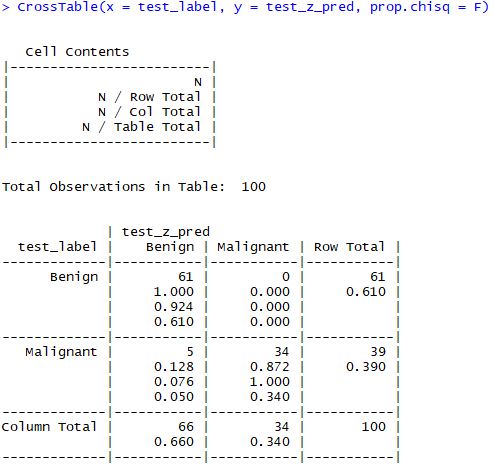
- 적용한 결과 정확도가 95%로 더 악화됬다는 사실을 알 수 있다.
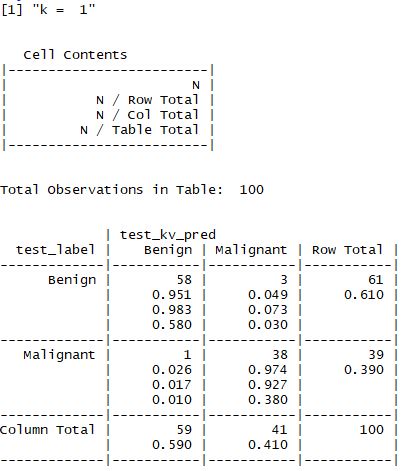
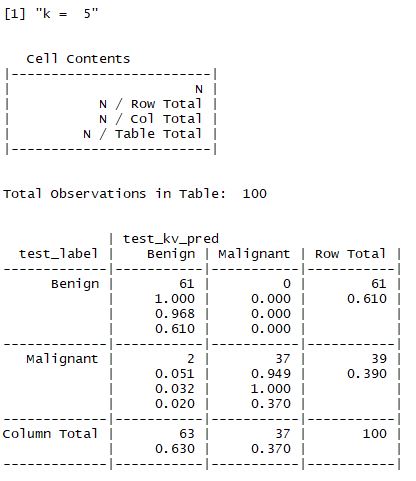
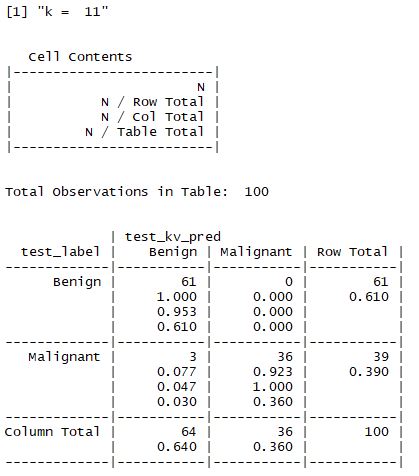
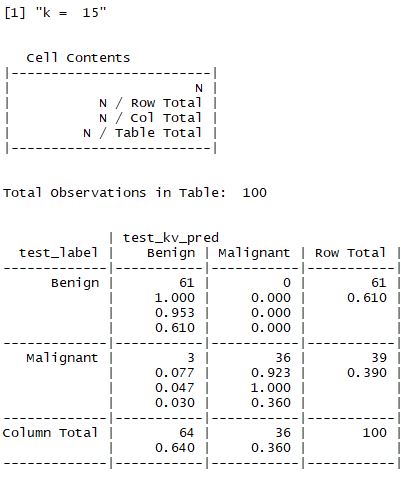
(2) k 값의 변화
- 앞서 설명한 것처럼 적절한 k값을 찾는 것이 중요하며 일반적으로는 3~10 사이의 값으로 지정한다고 했다.
- 해당 예제에서는 k값을 1,5,11,15,21,27 로 설정하여 값을 비교한다.
[결과]
① z - 표준화를 하지 않은 경우
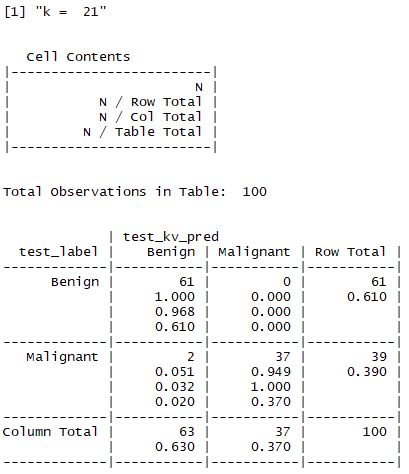
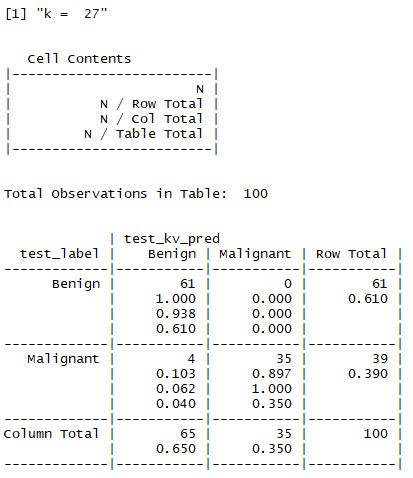
② z - 표준화를 한 경우
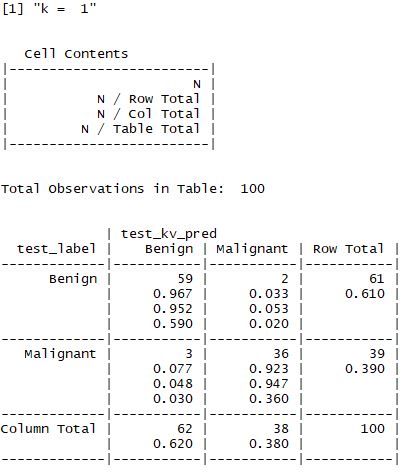
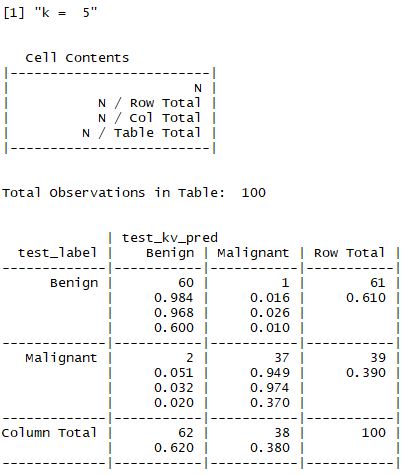
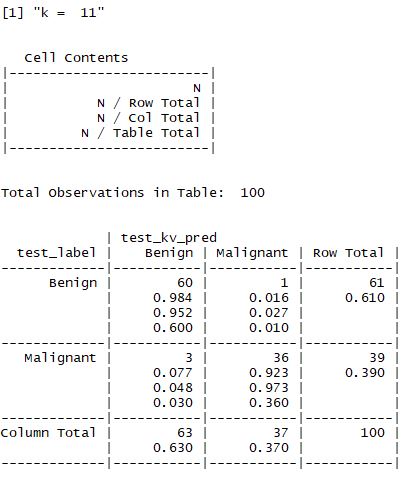
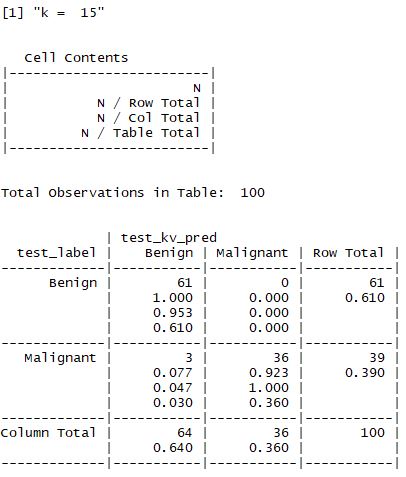
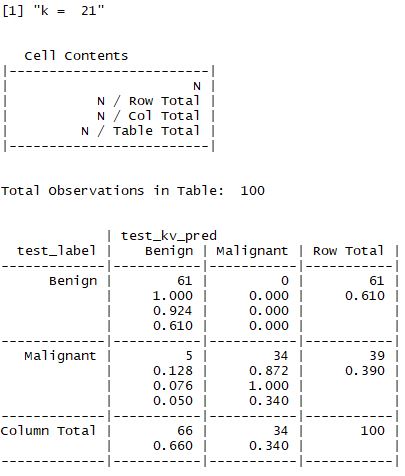
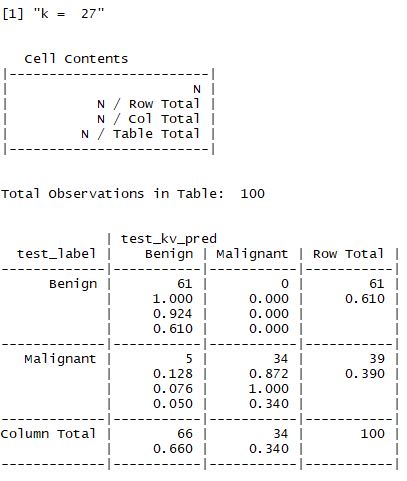
- 결과적으로 맨 처음 수행한 결과가 가장 좋으며 98%의 정확도를 갖는 모델이 선정된다.

댓글남기기